Fissaidee 3
| Site: | Federica Web Learning - LMS |
| Course: | Biologia di base |
| Unit: | Fissaidee 3 |
| Printed by: | Utente ospite |
| Date: | Tuesday, 26 August 2025, 7:24 PM |
1. La preparazione del DNA genomico
Il DNA genomico può essere preparato da qualunque campione biologico purchè contenga cellule fornite di nucleo. Per la specie Homo sapiens, il DNA può essere preparato ad esempio dal sangue (dalla sua componente linfo-monocitica), dalle cellule esfoliative buccali, dal bulbo pilifero, dagli amniociti ecc. Esistono varie metodiche che consentono di farlo, dalla classica estrazione con il Fenolo-Cloroformio, a più moderni sistemi con colonnine che contengono una membrana, generalmente di silice, in grado di legare e trattenere il DNA, fino ai sistemi automatizzati (estrattori automatici di DNA) basati sull’uso di biglie magnetiche che legano il DNA permettendone la separazione dalle altre componenti cellulari e quindi una ottimale purificazione.
2. La replicazione del DNA in vitro
Una delle funzioni principali del DNA, è quella di replicarsi, cioè di generare copie fedeli di sé stesso. Il meccanismo con cui avviene la replicazione è stato studiato negli anni immediatamente successivi al 1953 in cui è stato pubblicato su Nature il famoso articolo scientifico di Watson e Crick in cui descrissero la struttura molecolare a doppia elica del DNA. E già alla fine degli anni ’50 i ricercatori grazie agli studi di Kornberg erano in grado di far avvenire in una provetta da laboratorio la replicazione del DNA, processo indicato anche con “Sintesi del DNA” o “Duplicazione del DNA”.
Ma la vera rivoluzione per la genetica molecolare è stata la messa a punto di una metodica che consente di ottenere grandi quantità di DNA in una provetta di laboratorio. Tale metodica chiamata “Reazione a catena della polimerasi” o PCR, riproduce il meccanismo di replicazione del DNA che avviene nelle cellule. Tale metodica è stata inventata da Kary Mullis che per tale scoperta ebbe il premio Nobel nel 1993. La PCR permette di ottenere grandi quantità di una specifica sequenza di DNA in brevissimo tempo.
3. Il protocollo sperimentale della PCR
Per eseguire una PCR occorre preparare la reazione mettendo in una provetta i seguenti componenti: un buffer di reazione contenente Mg2+, una miscela dei 4 desossiribonucleotidi trifosfato, i due Primer specifici che fiancheggiano la regione da amplificare, l’enzima TaqPolimerasi e il DNA genomico da amplificare, che una volta denaturato fungerà da stampo.
La reazione di amplificazione consta di 3 step, ognuno realizzato ad una diversa temperatura, ripetuti ciclicamente un numero di volte stabilito dallo sperimentatore; in genere i cicli sono in numero variabile tra 20 e 40. I 3 step consistono in incubazioni per breve tempo (in genere 30-60 sec, in base alla lunghezza del segmento di DNA da amplificare) della reazione di amplificazione alle seguenti temperature:
| 1° step | 94-95°C per denaturare la doppia elica di DNA |
|---|---|
| 2° step | circa 55°C per permettere il legame dei primers al DNA stampo |
| 3° step | 72°C per la vera e propria sintesi del DNA |
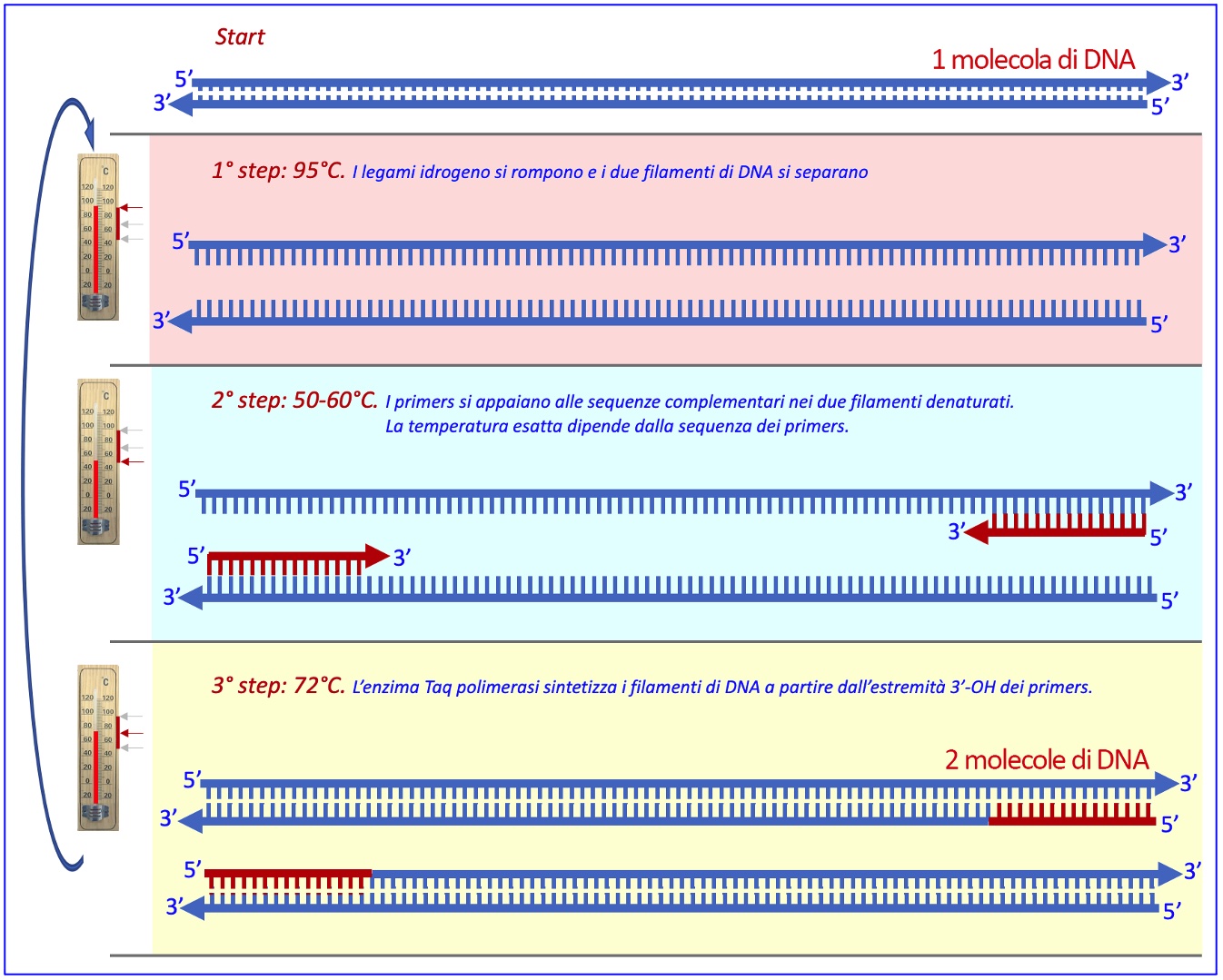
L’implementazione della metodica di PCR è stata resa possibile dalla scoperta della TaqPol, una DNA polimerasi termostabile estratta da un archeobatterio, Thermophilus acquaticus, che vive in condizioni di temperatura estreme. Questa DNA polimerasi a 95°C non si degrada, quindi la reazione può essere ripetuta ciclicamente molte volte.
4. Gli enzimi di restrizione
Sono chiamati “forbici del biologo molecolare”, perché permettono di tagliare il DNA a doppio filamento. I tagli avvengono in corrispondenza di siti di restrizione, specifici per ciascun enzima di restrizione. La maggior parte degli enzimi di restrizione sono isolati da batteri e tale derivazione è richiamata nei loro nomi, ad esempio BamHI è chiamato così perché è isolato da Bacillus amyloliquefaciens. I siti di restrizione, solitamente costituiti da 4 o 6 coppie di basi, possono essere tagliati in due modi diversi che generano estremità piatte o estremità adesive se il taglio produce brevi sporgenze a singolo filamento, complementari tra di loro.
Una delle caratteristiche peculiari degli enzimi di restrizione è l’elevatissima specificità con cui riconoscono e tagliano la sequenza corrispondente al sito di restrizione. È sufficiente un solo nucleotide differente per impedire all’enzima di tagliare il DNA. Questa caratteristica viene sfruttata in ambito biomedico per identificare punti di mutazione di singoli nucleotidi (SNP: Single Nucleotide Polymorphism) nel caso in cui la mutazione si verifichi all’interno di un sito di restrizione.
5. I polimorfismi della lunghezza dei frammenti di restrizione: RFLP
Il DNA amplificato contenente centinaia di migliaia di copie del tratto di DNA di interesse, può essere usato ad esempio per identificare polimorfismi genetici denominati RFLP (polimorfismi della lunghezza dei frammenti di restrizione). La tecnica utilizza enzimi di restrizione che tagliano esclusivamente in corrispondenza di specifiche sequenze e una mutazione (un polimorfismo genetico) può far sparire un sito di restrizione per un dato enzima oppure può farlo comparire. In ogni caso nell’analisi dei campioni su un gel di agarosio si può osservare un pattern di restrizione che risulta diverso nella lunghezza dei frammenti generati dalla restrizione tra i campioni che presentano la mutazione e quelli che non la presentano.
Una mutazione che fa scomparire un sito di restrizione determina la riduzione del numero dei frammenti di DNA ottenibili con una reazione di restrizione. Se invece la mutazione fa comparire un sito di restrizione, il numero di segmenti di DNA ottenibili con una reazione di restrizione aumenta.
6. L’elettroforesi
Un polimorfismo nella lunghezza dei frammenti di restrizione (RFLP) può essere rilevato mediante elettroforesi su gel di agarosio di un DNA prima amplificato mediante PCR e in seguito incubato con un enzima di restrizione. Il gel di agarosio in cui vengono caricati i campioni di DNA viene esposto ad un campo elettrico in modo che i segmenti di DNA presenti si muovano con una velocità inversamente proporzionale alla propria dimensione. La presenza, alla fine dell’elettroforesi, di un numero variabile di segmenti di DNA, di dimensioni specifiche (in relazione ad uno specifico RFLP), consente di stabilire il genotipo del campione analizzato. Il DNA all’interno del gel di agarosio e durante la corsa elettroforetica non è visibile. La visualizzazione avviene dopo esposizione del gel, con il DNA all’interno, a una sorgente di radiazioni a specifiche lunghezze d’onda che eccitano una molecola (il SYBR Green) che emette una luce fluorescente visibile. Il SYBR Green emette la fluorescenza solo se presente all’interno della doppia elica del DNA, quindi la sua visualizzazione identifica, la posizione del DNA lungo il gel.
7. L’identificazione dei genotipi mediante RFLP
L’RFLP non è l’unica procedura che permette attualmente la identificazione sperimentale di un genotipo. I notevoli avanzamenti delle biotecnologie hanno consentito oggi lo sviluppo di nuove metodiche come l’NGS (Next Generation Sequencing) che permette di genotipizzare simultaneamente anche decine o centinaia di geni.
La metodica degli RFLP è comunque oggi usata e preferita in molti contesti di ricerca e di diagnosi, per esempio per diagnosticare lo stato di portatore sano di Anemia Falciforme. È sufficiente effettuare una amplificazione mediante PCR del DNA genomico con primer che amplificano la regione contenente la mutazione. Effettuare una digestione enzimatica con un enzima che taglia/non taglia in base alla presenza/assenza della mutazione e osservare il risultato mediante una elettroforesi in gel di agarosio.