3. Il protocollo sperimentale della PCR
Per eseguire una PCR occorre preparare la reazione mettendo in una provetta i seguenti componenti: un buffer di reazione contenente Mg2+, una miscela dei 4 desossiribonucleotidi trifosfato, i due Primer specifici che fiancheggiano la regione da amplificare, l’enzima TaqPolimerasi e il DNA genomico da amplificare, che una volta denaturato fungerà da stampo.
La reazione di amplificazione consta di 3 step, ognuno realizzato ad una diversa temperatura, ripetuti ciclicamente un numero di volte stabilito dallo sperimentatore; in genere i cicli sono in numero variabile tra 20 e 40. I 3 step consistono in incubazioni per breve tempo (in genere 30-60 sec, in base alla lunghezza del segmento di DNA da amplificare) della reazione di amplificazione alle seguenti temperature:
| 1° step | 94-95°C per denaturare la doppia elica di DNA |
|---|---|
| 2° step | circa 55°C per permettere il legame dei primers al DNA stampo |
| 3° step | 72°C per la vera e propria sintesi del DNA |
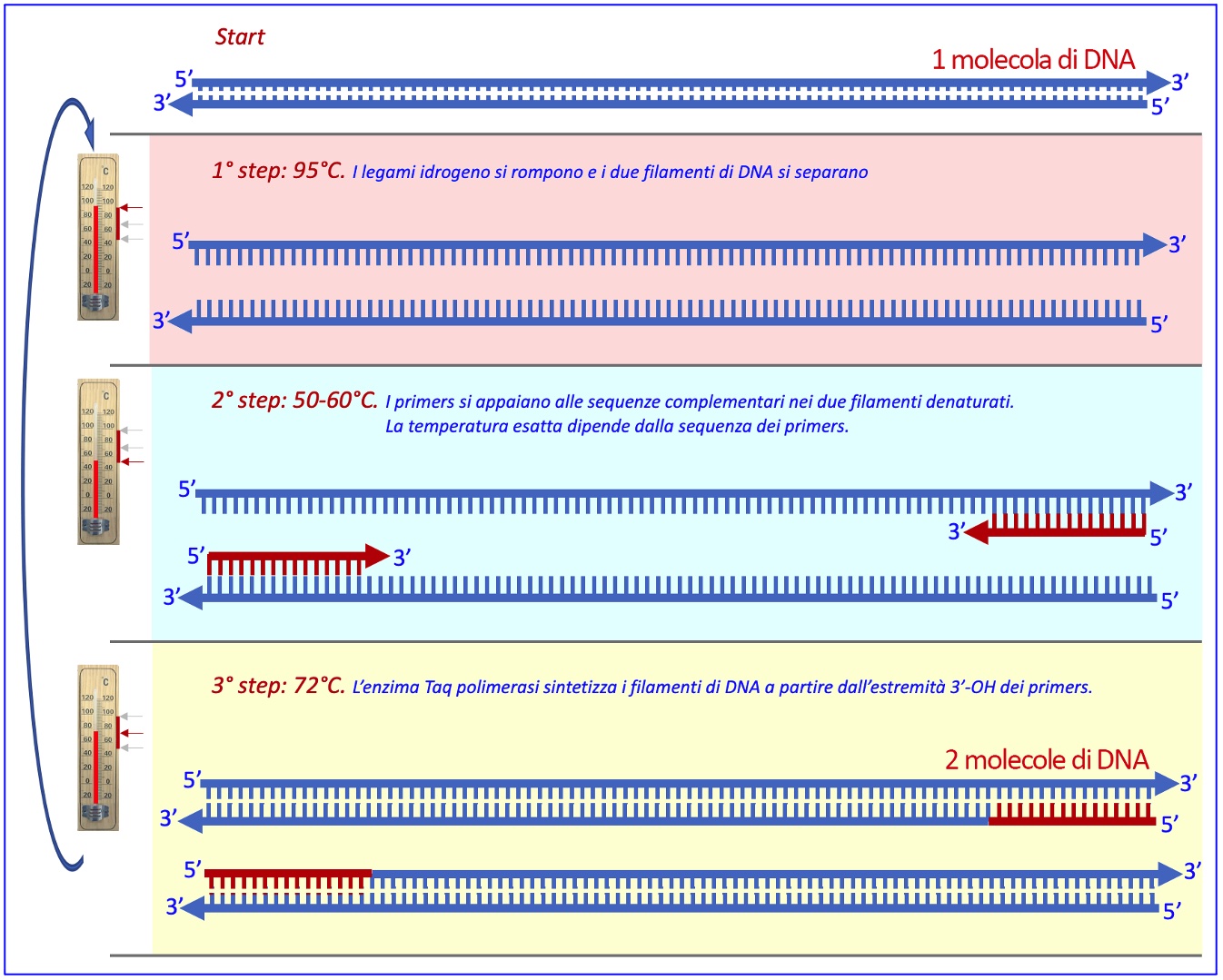
L’implementazione della metodica di PCR è stata resa possibile dalla scoperta della TaqPol, una DNA polimerasi termostabile estratta da un archeobatterio, Thermophilus acquaticus, che vive in condizioni di temperatura estreme. Questa DNA polimerasi a 95°C non si degrada, quindi la reazione può essere ripetuta ciclicamente molte volte.
